- 品牌
- 上海云序生物
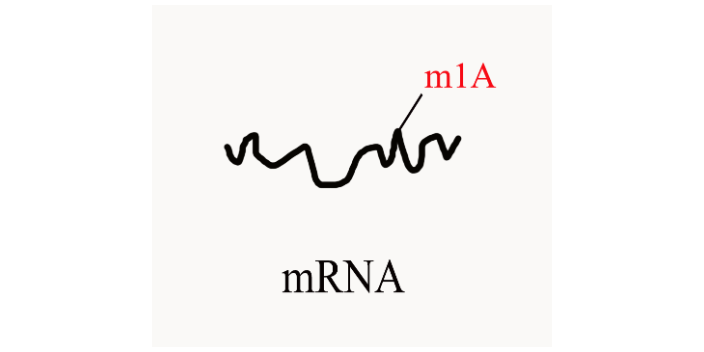
m1A RNA甲基化是一类新发现的RNA甲基化,即RNA分子腺嘌呤第1位氮原子上的甲基化修饰(N1-methyladenosine,m1A)。研究表明,m1A是真核生物tRNA和rRNA丰度很高的一种转录后修饰,近期研究也表明m1A修饰可调控mRNA翻译。m1A修饰作为一类新型RNA甲基化,其功能和机制都亟待挖掘。 与m6A RNA检测方式一致,针对m1A修饰也采用MeRIP-seq技术,抗体富集的技术原理如下:将m1A RNA甲基化特异性抗体与被随机打断的RNA的片段进行共孵育,抓取有甲基化修饰的片段进行测序;同时需要平行测序一个对照(Input)样本,对照样本为未进行IP反应的RAN片段,对照样本用于消除非特异性抓取甲基化片段的背景。2’-O-RNA甲基化修饰在mRNA、tRNA、rRNA、miRNA等分子上分布。山西甲基化研究
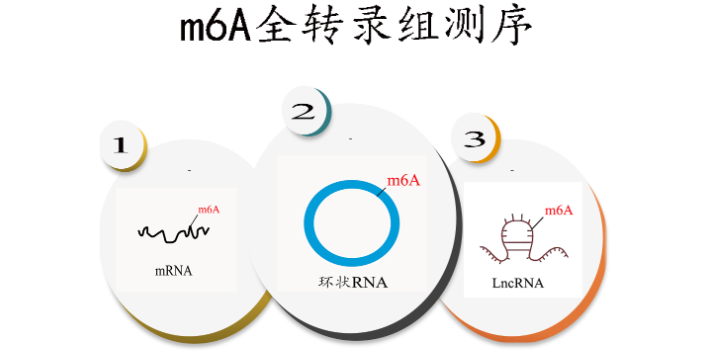
在这篇文章中,发现番茄果实成熟过程中,mRNA m6A甲基化表现出与DNA甲基化相似的动态变化。RNA甲基组分析表明,m6A甲基化是番茄果实mRNA中普遍存在的修饰,且m6A位点富集于终止密码子周围和3'UTR区。在具有DNA超甲基化的成熟缺陷表观突变体无色非成熟(Cnr)的果实中,有1100多个转录本显示m6A水平升高,而只有134个转录本显示m6A水平降低,表明m6A的整体水平升高。m6A沉积通常与转录本丰度呈负相关。云序优势 一站式服务: 客户只需提供细胞、组织或RNA,云序生物为您完成从MeRIP富集,文库制备,上机测序到数据分析整套服务流程。 rDNA甲基化筛选m5C RNA是近年来发现的一类在tRNA及rRNA高丰度存在的甲基化修饰。
m6A修饰在植物中是必不可少的。在这里,作者证明了在水稻中表达转基因人RNA去甲基化酶FTO可以使温室条件下的粮食产量增加3倍以上。在田间试验中,转基因FTO在水稻和马铃薯中的表达使产量和生物量增加了约50%。此外,FTO的存在促进了根分生组织细胞增殖和分蘖芽的形成,提高了光合效率和抗旱性,但对成熟细胞大小、茎分生组织细胞增殖、根直径、株高或倍性没有影响。FTO介导了植物RNA中大量的m6A去甲基化(在poly(A) RNA中约7%的去甲基化,在非核糖体核RNA中m6A下降约35%),诱导染色质开放和转录激huo。因此,调控植物RNA m6A甲基化是一种很有前景的策略,可明显提高植物的生长和作物产量。
样本要求 1)样品类型:细胞、新鲜组织或RNA 样品。 2)样品量: 细胞:2×107 组织:500 mg-1 g RNA:30 - 300 μg,浓度不低于100 ng/μL 3)RNA 质量要求: OD260/280:1.8~2.1 浓度≥ 100 ng/μl RNA 无明显降解(28S:18S ≥ 1.5 或者RIN ≥ 7) 4)样品保存: 新鲜组织可用RNA保护剂处理或液氮冻存后,-80℃保存。 细胞样品,离心收集细胞沉淀后用PBS洗2遍,直接-80℃保存;RNA 样品可溶于乙醇或RNA-free 的超纯水中,-80℃保存。样品保存 Note:样品保存期间避免反复冻融。 5)样品运输:样品置于1.5 ml Eppendorf管或冻存管中,封口膜封好,装在塑料袋中,干冰运输。对m6A RNA甲基化,目前较流行的检测手段为m6A-Seq技术。
案例2:2’-O-RNA甲基化酶FTSJ3协助HIV病 毒逃避宿主细胞免疫识别机制 原文:FTSJ3 is an RNA 2’-O-methyltransferase recruited by HIV to avoid innate immune sensing 期刊:Nature 影响因子:41.6 外国研究人员首先借助蛋白互作实验证实TRBP蛋白能够在不依赖于DICER酶的作用下与FTSJ3结合,形成复合物,因此推测,FTSJ3是一种2'-O-甲基化转移酶。接着,体外和体内实验表明FTSJ3就是一种通过TRBP被招募到HIV RNA上的2'-O-甲基化转移酶。通过优化的2’-O-RNA甲基化测序手段,证实在HIV病 毒遗传信息的特定位置上存在依赖于FTSJ3的2'-O-甲基化修饰。综上,该研究证实TRBP-FTSJ3复合物通过招募到HIV病 毒RNA发生2'-O-甲基化从而解释了HIV病 毒逃避宿主细胞先天免疫识别的机制。云序生物自2018年推出了超微量RNA甲基化测序。江苏甲基化研究
适用于m6A RNA甲基化谱研究,快速筛选m6A RNA甲基化靶基因。山西甲基化研究
案例1:m5C RNA甲基化调控mRNA出核新机制 原文:5-methylcytosine promotes mRNA export — NSUN2 as the methyltransferase and ALYREF as an m5C reader 期刊:Cell Research 影响因子:15.60 中科院汪海林等研究团队揭示了m5C修饰在mRNA的分布图谱规律及其对调控mRNA出核作用新机制。研究人员建立了改进的RNA m5C单碱基分辨率高通量测序与生物信息分析技术,以此揭示mRNA m5C的分布规律,并绘制了精细的m5C修饰图谱,发现m5C在mRNA的翻译起始位点下游有明显富集,并且主要分布于CG富集区域。通过分析对比人和小鼠不同组织,发现m5C在mRNA上的分布特征在哺乳动物中十分保守,而在不同组织中修饰的基因具有特异性。研究团队同时发现,在小鼠睾丸发育过程中,动态的m5C修饰基因明显富集于精子发育相关功能,提示m5C修饰参与生殖发育调控。山西甲基化研究
- 吉林DNA甲基化 2023-08-07
- 河北miRNA甲基化 2023-08-07
- 内蒙古tRNA甲基化 2023-08-07
- 宁夏超微量RNA甲基化 2023-08-07
- 甲基化表达 2023-08-07
- 云南甲基化研究 2023-08-07
- 外泌体甲基化过程 2023-08-07
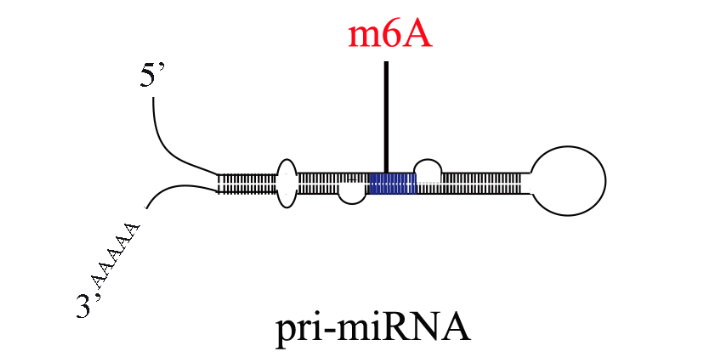
- 四川pri-miRNA甲基化 2023-08-07
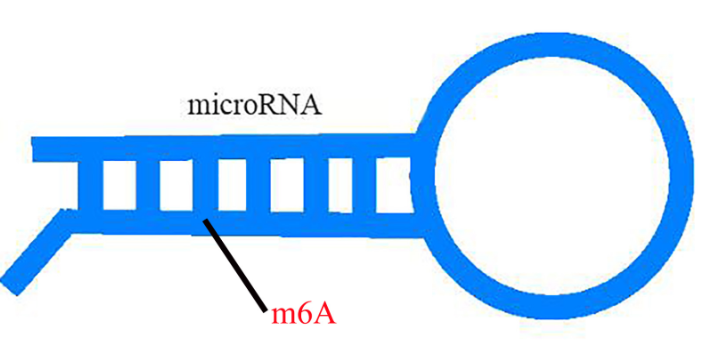
- 辽宁microRNA甲基化 2023-08-06
- DNA甲基化调控 2023-08-06
- 陕西microRNA甲基化 2023-08-06
- 西藏甲基化测序 2023-08-06