Q:ChIP-Seq和ChIP-qPCR有何异同?A:染色质免疫共沉淀(ChIP)所获得的DNA产物,在ChIP-Seq中通过高通量测序的方法,在全基因组范围内寻找目的蛋白(转录因子、修饰组蛋白)的DNA结合位点片段信息;ChIP-qPCR需要预设待测的目的序列,针对目的序列设计引物,以验证该序列是否同实验蛋白结合互作。
Q:染色质片段大小在哪个范围比较合适?A:对于ChIP-seq,片段在200-500bp左右是合适范围;对于ChIP-qPCR,片段在200-800bp左右适宜。
Q:植物样本处理和动物组织/细胞有何区别?A:植物组织由于细胞壁、气腔等结构的存在,会给交联缓冲液的作用带来困难,因此相对于动物组织/细胞来说,往往需要在抽真空条件下进行交联,而该步奏是一个需要经验及优化的过程。
Q:ChIP-Seq中的测序DNA样本需要多少产量?A:通常是≥10ng。
Q:ChIP风险如果判断A:ChIP实验以标签来判断实验风险,重组标签的转录因子>内源转录因子>组蛋白;当以重组蛋白作为靶蛋白时,重组蛋白同内源蛋白可能存在结合活性、结合位点差异;以标签抗体进行ChIP时、染色质结合位点本身会被内源蛋白竞争,这些都会影响到ChIP过程的特异性捕获效率。 染色质免疫沉淀(Chromatin Immunoprecipitation,ChIP)是研究体内蛋白质与DNA相互作用的一种技术。chromatin免疫沉淀ChIP-Sequencing检测

转录因子机制研究是一个复杂的过程,涉及多个步骤和技术。转录因子机制研究建议(二)。执行实验:按照实验计划进行操作,记录实验过程和结果。确保实验操作的准确性和可重复性。数据分析:使用适当的统计方法和软件对实验数据进行处理和分析。将结果与已知数据进行比较,并解释发现。验证和扩展研究:对初步结果进行验证,并通过进一步实验来扩展研究。这可能包括使用不同的细胞类型、条件或技术来验证发现。撰写和发表研究成果。转录因子机制研究确保遵循科学的研究方法和规范,持续学习和更新知识,以提高研究的质量和影响力。DNA蛋白互作检测ChIP通过ChIP-qPCR分析转录因子结合位点的富集程度,为转录因子结合位点的功能研究提供实验依据。

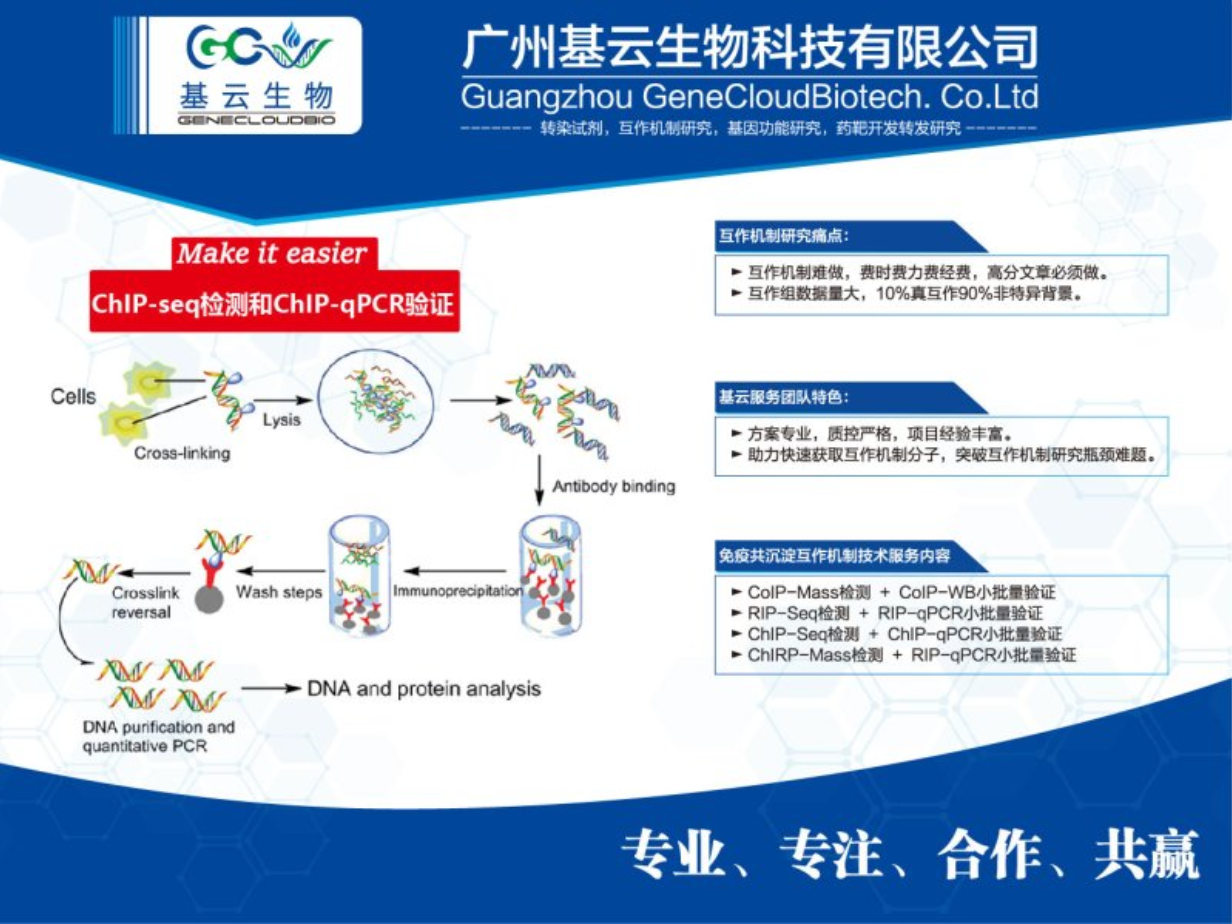
ChIP实验关键步骤1)细胞的准备及固定细胞在培养皿上生长到80%~90%。当做实验时,可以同时准备两个培养皿,一个用于预测细胞数量(细胞量为1E5),另外一个用于优化超声条件。2)染色质超声断裂加入SDS裂解溶液重悬沉淀,在冰上放置10min,每隔1min颠倒摇动离心管。SDS能裂解细胞膜和核膜,使固定染色质释放出来,这样有利于超声。3)免疫沉淀蛋白和DNA复合物所有染色质免疫沉淀步骤都必须低温(冰上或4℃)操作。使用ChIP稀释溶液把超声染色质液体稀释10倍,这样有利于免疫沉淀反应。为了减少非特异性结合蛋白背景,往2mL超声稀释液中加入20μL蛋白A/G琼脂糖珠(Protein A/G Agarose Beads),在4℃摇床轻柔摇动1h。4)洗脱、解交联从这一步开始,所有操作都在室温进行。在洗脱步骤完成后吸取洗脱上清时要格外注意,防止吸走微珠而造成样品污染。同时要注意每个样品管吸取等量的上清,防止造成样品间误差。5)DNA纯化DNA纯化可以通过酚/氯仿抽提或者通过硅胶柱纯化。6)鉴定一旦完成了DNA纯化,便可进行多种下游分析,包括ChIP-PCR、ChIP-qPCR、ChIP-芯片和ChIP-seq。
ChIP-seq实验虽然是一种强大的研究蛋白质与DNA相互作用的技术,但也存在一些缺点。首先,ChIP-seq实验需要大量的起始材料,通常需要数百万个细胞,这对于某些稀有或难以培养的细胞类型来说是一个挑战。其次,ChIP-seq实验的过程相对复杂,需要经过多个步骤,包括细胞交联、染色质片段化、免疫沉淀、文库构建和高通量测序等。每个步骤都可能引入误差或偏差,需要仔细优化和控制实验条件。此外,ChIP-seq实验的结果受到抗体特异性和亲和力的影响。如果使用的抗体质量不高或与目标蛋白质的结合不够特异和紧密,可能会导致结果的假阳性或假阴性。另外,ChIP-seq实验的数据分析也是一个挑战。由于测序产生的数据量庞大,需要专业的生物信息学知识和技能进行有效的数据处理和解读。同时,ChIP-seq实验的结果通常需要在基因组注释、转录因子结合位点数据库等多个层面进行整合和验证,以确保结果的准确性和可靠性。综上所述,尽管ChIP-seq实验是一种强大的技术,但在实际应用中需要考虑其局限性,并仔细设计实验方案、优化实验条件、选择合适的抗体和进行有效的数据分析。作为新手开展ChIP实验,需要注意哪些问题。
使用ChIP-seq快速确定下游靶标涉及多个关键步骤:首先,进行ChIP实验以富集与目标蛋白(如转录因子)结合的DNA片段。在这一步中,确保使用高质量的抗体以特异性地捕获目标蛋白与DNA的复合物。接着,将富集的DNA片段进行高通量测序。测序产生的数据将提供全基因组范围内目标蛋白的结合位点信息。然后,对测序数据进行生物信息学分析。这包括将测序读段比对到参考基因组上,识别并注释峰值区域,这些峰值区域表示目标蛋白与DNA的潜在结合位点。接下来,分析峰值区域在基因组中的分布,以确定下游靶标。特别关注那些位于基因启动子、增强子等调控区域的峰值,因为这些区域通常与基因表达调控密切相关。此外,还可以整合其他组学数据(如转录组学、表观遗传学数据等),以进一步验证和解释目标蛋白与下游靶标之间的调控关系。另外,通过实验验证(如qPCR、基因敲除或过表达等)来确认下游靶标的功能和调控作用。综上所述,通过ChIP-seq实验结合生物信息学分析和实验验证,可以快速而准确地确定下游靶标,并揭示目标蛋白在基因表达调控网络中的作用机制。ChIP-qPCR实验虽然是一种有效的研究蛋白质与DNA相互作用的方法,但也存在一些缺点。四川ChIP-qPCR检测
ChIP-seq(染色质免疫沉淀测序)是一种强大的实验技术,广泛应用于多个生物学领域。chromatin免疫沉淀ChIP-Sequencing检测
ChIP-seq与ChIP-qPCR在实验原理和应用方面存在一些相同点。首先,它们的实验原理都基于染色质免疫沉淀(ChIP),这是一种用于研究蛋白质与DNA相互作用的技术。它们都通过特异性抗体与目标蛋白质结合,形成免疫复合物,从而富集与特定蛋白质结合的DNA片段。其次,ChIP-seq与ChIP-qPCR在实验步骤上也有相似之处。它们都需要进行交联、裂解、染色质片段化、免疫沉淀和解交联等步骤。在这些步骤中,它们都利用特异性抗体来捕获目标蛋白质与DNA的复合物,并通过洗涤去除非特异性结合,得到富集的DNA片段。ChIP-seq与ChIP-qPCR都应用于研究蛋白质在基因组上的结合情况。通过这两种技术,我们可以了解转录因子、组蛋白修饰等蛋白质在全基因组范围内的结合位点,从而揭示基因表达调控的机制。不过,它们也存在不同之处。ChIP-seq结合了高通量测序技术,可以在全基因组范围内分析蛋白质与DNA的相互作用,提供更高分辨率的结合位点信息。而ChIP-qPCR则侧重于对特定基因或基因区域的定量分析,具有更高的灵敏度和特异性。因此,在实际应用中,我们可以根据研究需求选择合适的技术方法。chromatin免疫沉淀ChIP-Sequencing检测