- 品牌
- 上海云序生物
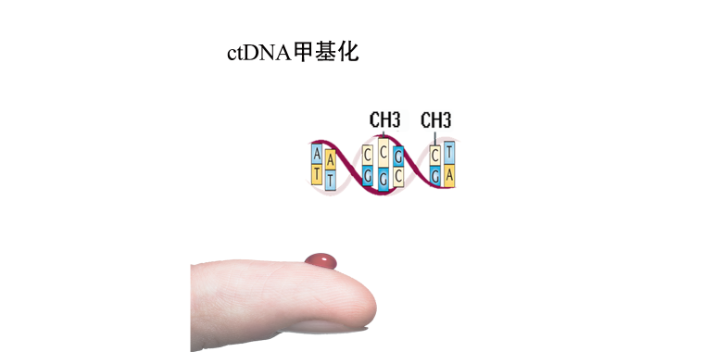
研究表明,RNAm6A修饰影响mRNA的转录、定位、翻译、稳定性、剪接和核输出。然而,转录组m6A谱及其在白菜热胁迫中的潜在生物学作用尚缺乏足够的资料。通过MeRIP-seq获得白菜中RNA m6A修饰的first转录组全谱。同时,通过分析Input测序数据获得转录组数据。发现在正常组(CK)和热应激组(T43)中鉴定出11252个m6A共有峰和9729个含有m6A的共有基因。且CK组和T43组中,m6A峰均在3’UTR区高度富集。大约80%的基因有一个m6A位点。m6A峰的共识基序为AAACCV (V: U/A/G)。此外,关联分析发现m6A的甲基化程度与转录水平存在一定的相关性,说明m6A在基因表达中起一定的调控作用。ctDNA 的 5mC 甲基化修饰研究仍然是一个未被充分探索的领域。贵州6mADNA甲基化
Nature | miR-1的位置特异性氧化导致心脏肥大 发表时间:2020年8月5日 影响因子:42.778 Nature上发表的来自韩国高丽大学Sung Wook团队的一篇研究在氧化还原相关疾病心肌肥大的大鼠模型中对氧化的miRNA进行了特异性测序研究。 作者在氧化还原相关疾病心肌肥大的大鼠模型中对氧化的microRNA(miRNA)进行了特异性测序。结果发现8-氧代鸟嘌呤2(o8G)修饰是选择性地在miRNA的特殊区域(位置2-8)产生的,并通过o8G-A碱基互补配对来调节其他mRNA。用肾上腺素能激动剂医治后主要在miR-1(7o8G-miR-1)的第7位诱导了o8G氧化修饰。单独引入的7o8G-miR-1或7U-miR-1(其中7位的G被U取代)足以引起小鼠心脏肥大,o8G-miR-1的mRNA靶基因在受影响的表型中起作用,反之对小鼠心肌细胞7o8G-miR-1的特异性抑制可减轻心脏肥大。o8G-miR-1也与心肌病患者有关。本研究表明miRNA的位置特异性氧化可以作为表观转录机制来协调病理生理学氧化还原介导的基因表达,为研究氧化修饰在病理生理学中的作用提供了新的思路。浙江850KDNA甲基化RNA m6A是真核生物丰富和调节遗传信息的一种保守的转录后机制。
云序生物对O8G RNA氧化化检测手段为O8G-IP-Seq技术,技术原理如下: 将O8G特异性抗体与被随机打断的RNA的片段进行共孵育,抓取有O8G修饰的片段进行测序;同时需要平行测序一个对照(Input)样本,对照样本只含有打断的RNA的片段,并不添加O8G特异性抗体与其共孵育。对照样本用于消除非特异性抓取的O8G片段的背景。对比免疫共沉淀(IP)样本和对照样本(Input)中的序列片段,将O8G氧化修饰位点定位到转录组上,并根据RNA-seq数据,计算样本中O8G氧化程度及对RNA表达水平的影响。
m5C RNA是近年来发现的一类在tRNA及rRNA高丰度存在的甲基化修饰。利用高通量测序手段验证了非编码RNA以及部分mRNA中m5C存在,但是在不同物种、不同组织中m5C修饰分布图谱尚没有系统性报道。云序生物率先开展m5C RNA甲基化测序服务,采用经典重亚硫 酸盐处理的方式进行测序。在全转录范围内及tRNA水平查看基因m5C甲基化修饰水平。 通过高通量测序和生物信息分析,识别甲基化富集的基因组区域。 富集峰识别后,得到的是一堆基因组位置信息,通过生物信息分析利用邻近基因对富集峰进行注释,并根据峰中点相对于已知基因的位置,将富集峰为启动子峰、上游峰、内含子峰、外显子峰、基因间峰。 研究表明,m7G RNA甲基化修饰存在于各类分子中。
样品类型 细胞、组织或RNA,其他样本类型请电询。 样品量 组织:100 mg – 1 g 细胞:2× 107 个以上 RNA:30-300 μg (OD260/280:1.6-2.3;RNA无明显降解28S:18S>1.5或RIN>7) 样品保存与运输 样品保存:细胞样品或新鲜组织块(切成 5-10 mg 的小块),可用 TRIzol 或 RNA 保护剂处理,液氮冻存后,-80℃ 保存, RNA 样品可溶于乙醇或 RNA-free 的超纯水中,-80℃ 保存,样品保存期间避免反复冻融。 样品运输:样品置于 1.5 mL 离心管中,封口膜封好,埋在盛有干冰的泡沫盒中运输。目前对m6A RNA流行检测手段为MeRIP-Seq技术。西藏tRNA甲基化
RNA 修饰在基因表达调控中扮演着非常重要的角色。贵州6mADNA甲基化
案例2: m5C RNA甲基化基因组分布图谱 原文:Distinct 5-methylcytosine profiles in poly(A) RNA from mouse embryonic stem cells and brain 期刊:Genome Biology 影响因子:13.21 近期刊登了解析RNA m5C在不同组织细胞的分布图谱相关文章。研究人员选取小鼠胚胎干细胞(ESC)和脑组织(n=3)作为实验样本,利用m5C RNA甲基化测序技术对两组样本中的总RNA和胞核RNA进行检测。结果表明,ESC中总RNA 5mC的分布频率比脑组织的分布频率更高。研究人员将m5C位点和不同的基因组区域(CDS, 5’-UTR, 3’-UTR等)进行了关联分析。结果表明,总RNA中m5C位点更倾向于分布在转录起始位点。脑组织和ESC中,3’-UTR区m5C的分布区别很大,这暗示着3’-UTR区的RNA m5C修饰可能与细胞功能和细胞类型密切相关。贵州6mADNA甲基化
- 吉林DNA甲基化 2023-08-07
- 河北miRNA甲基化 2023-08-07
- 内蒙古tRNA甲基化 2023-08-07
- 宁夏超微量RNA甲基化 2023-08-07
- 甲基化表达 2023-08-07
- 云南甲基化研究 2023-08-07
- 外泌体甲基化过程 2023-08-07
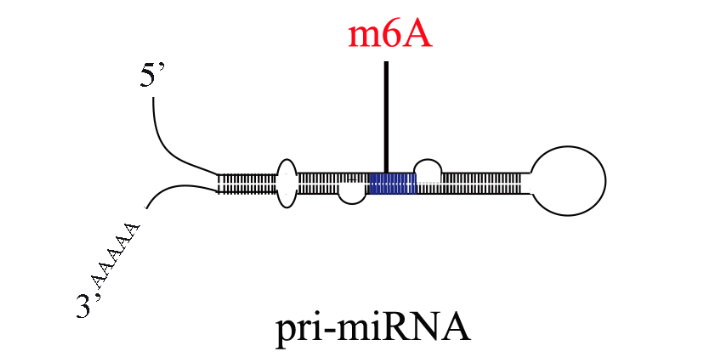
- 四川pri-miRNA甲基化 2023-08-07
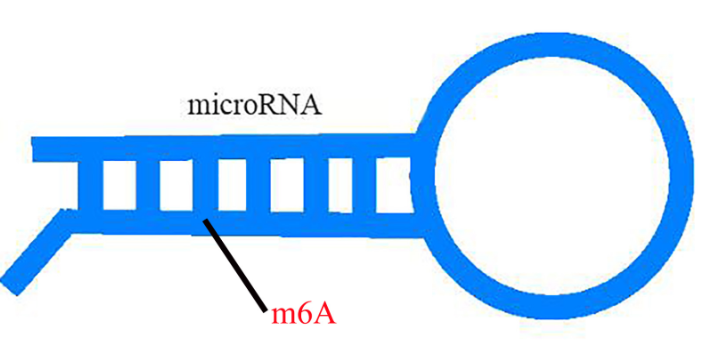
- 辽宁microRNA甲基化 2023-08-06
- DNA甲基化调控 2023-08-06
- 陕西microRNA甲基化 2023-08-06
- 西藏甲基化测序 2023-08-06